numpy.polynomial.legendre.legfit¶
-
numpy.polynomial.legendre.legfit(x, y, deg, rcond=None, full=False, w=None)[source]¶ Legendre系列的最小二乘法拟合数据。
返回度数deg的系数,该系数是在点x给出的数据值y的最小二乘拟合。如果y是1-D,则返回的系数也将是1-D。如果y是2-D多重拟合,对于y的每一列进行一次,并且所得到的系数存储在2-D返回的相应列中。拟合的多项式是形式
其中n是deg。
参数: x:array_like,shape(M,)
M个采样点
(x [i], y [i])的x坐标。y:array_like,shape(M,)或(M,K)
y坐标。通过传递每列包含一个数据集的2D阵列,可以一次拟合共享相同x坐标的样本点的若干数据集。
deg:int或1-D array_like
拟合多项式的度(s)。如果deg是单个整数,则包括deg项的所有项包括在拟合中。对于Numpy版本> = 1.11,可以使用指定要包括的术语的度数的整数列表。
rcond:float,可选
相对条件编号。相对于最大奇异值小于该值的奇异值将被忽略。默认值为len(x)* eps,其中eps是float类型的相对精度,在大多数情况下约为2e-16。
full:bool,可选
开关确定返回值的性质。当它为False(默认值)时,只返回系数,当来自奇异值分解的True诊断信息也返回时。
w:array_like,shape(M,),可选
重量。如果不是无,则通过w [i]加权每个点
(x[i],y[i])对拟合的贡献。理想地,选择权重使得乘积w[i]*y[i]的误差都具有相同的方差。默认值为“无”。版本1.5.0中的新功能。
返回: coef:ndarray,shape(M,)或(M,K)
勒让德系数从低到高排序。如果y是2-D,则y的列k中的数据的系数在列k中。如果deg被指定为列表,则在返回的coef中将未包括在拟合中的项的系数设置为等于零。
[residuals,rank,singular_values,rcond]:list
只有full = True时,才会返回这些值
resid – sum of squared residuals of the least squares fit rank – the numerical rank of the scaled Vandermonde matrix sv – singular values of the scaled Vandermonde matrix rcond – value of rcond.
有关详细信息,请参阅linalg.lstsq。
警告: RankWarning
最小二乘法拟合中的系数矩阵的秩是不足的。只有在满 = False时,才会发出警告。警告可以通过关闭
>>> import warnings >>> warnings.simplefilter('ignore', RankWarning)
也可以看看
chebfit,polyfit,lagfit,hermfit,hermefitlegval- 评估Legendre系列。
legvander- Vandermonde矩阵的Legendre系列。
legweight- Legendre权重函数(= 1)。
linalg.lstsq- 从矩阵计算最小二乘拟合。
scipy.interpolate.UnivariateSpline- 计算样条拟合。
笔记
解决方案是Legendre系列p的系数,其使加权平方误差的和最小化
其中
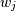 是权重。这个问题通过设置为(通常)过度确定的矩阵方程来解决
是权重。这个问题通过设置为(通常)过度确定的矩阵方程来解决其中V是x的加权伪Vandermonde矩阵,c是要求解的系数,w权重和y是观察值。然后使用V的奇异值分解来求解该方程。
如果V的一些奇异值如此小以至于被忽略,则将发出RankWarning。这意味着可能不良地确定系数值。使用较低的顺序通常会摆脱警告。rcond参数也可以设置为小于其默认值的值,但是所得到的拟合可能是假的并且具有来自舍入误差的较大贡献。
使用Legendre系列的适配器通常比使用幂级数的适配器更好,但很大程度上取决于采样点的分布和数据的平滑度。如果拟合的质量不够,花键可能是一个好的选择。
参考文献
[R64] 维基百科,“曲线拟合”,http://en.wikipedia.org/wiki/Curve_fitting